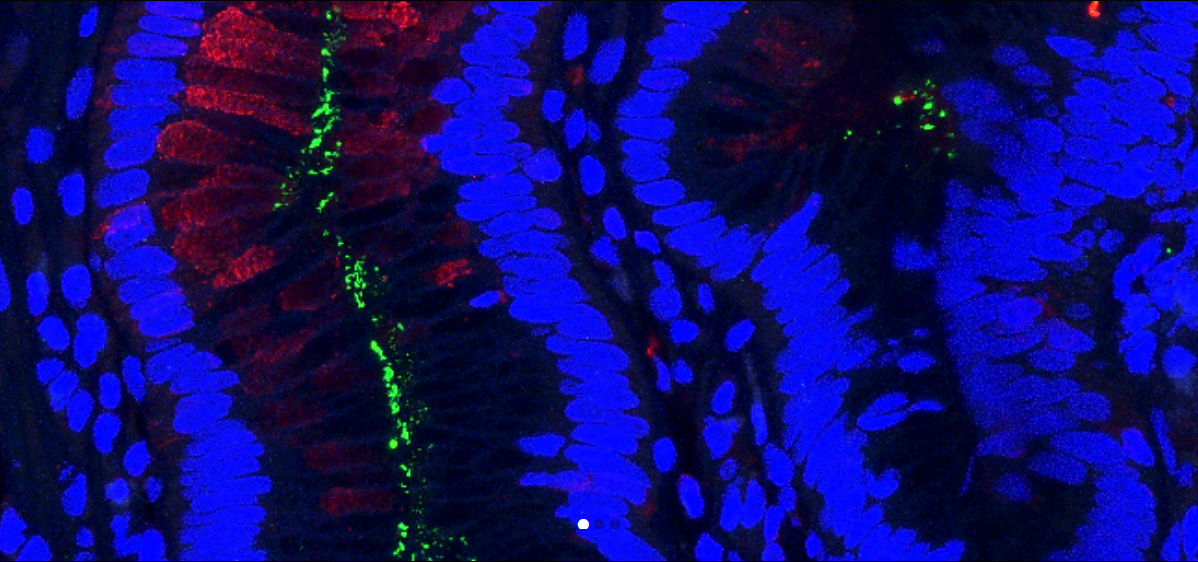
In der Medizinischen Biotechnologie, forschen wir an neuen Ansätzen um die medizinische Praxis der Zukunft zu gestalten. An unserem Fachbereich steht der Mensch im Mittelpunkt. Wir verzichten dewegen auf Tierversuche und widmen uns ganz dem Studium humaner Zellen und Gewebe, dies verwirklichen wir unteranderem durch innovative "state of the art" Kulturstrategien. Dabei setzen wir schon seit längerem auf 3D Kultivierung, wie zum Beispiel der Chip-Basierten Kultvierung von Zellen sowie der Anwendung von Organoid-Modellen, die bei uns zur Nachbildung des Kochenmarks bzw. des Magendarmtrakts verwendet werden
Ziel dabei ist es die tatsächlichen Abläufe im menschlichen Körper möglichst lebensecht (In vivo) abzubilden um ein tieferes Verständniss über die Mechnanismen zu erhalten, welche über Gesundheit und Krankheit entscheiden. Wir sind davon überzeugt, dass unsere Forschungszweig richtungsweisende Erkenntnisse liefern wird, von der in Zukunft viele Menschen profitieren werden.
Weitere Informationen über unsere Forschung und Aktuelles aus dem Labor, finden Ihr hier auf unserer Website oder auf BartfeldLab.com.
Neuigkeiten über unser Fachgebiet erhaltet ihr auch auf unserer Facebook-Seite.